インプット(input)サンプルについて
ChIP-qPCR法によってDNAを定量する際、実験系によって適切なコントロールを置くことが不可欠です。
ChIPでは抗体によって免疫沈降する手順がありますが、免疫沈降実験では免疫沈降させる前のサンプルをインプットとして、免疫沈降したサンプルと共に泳動・ウェスタンブロットを行います。これには使用したサンプルにどのくらいの量の目的の因子が入っていたかを示す意義があります。
ChIP-qPCRでは目的タンパク質と共沈したDNAを定量するため、同様にクロマチンの断片化後、免疫沈降の手順を行わないサンプルをインプットとして脱クロスリンク・精製し、インプットに含まれるDNA量に対する、免疫沈降後のDNAの回収率を定量値として用います。
サンプル間の差は免疫沈降したサンプルの、インプットサンプルに対する比を比較することになります。
例を使って説明します
100 ug / mlのクロマチンDNAサンプルがあったとします。
インプットとして100 μlのサンプルを取っておき、ChIPに1 mlを使います。
ChIP後、インプットサンプルとChIPしたサンプル両方を脱クロスリンクして沈殿し、qPCRを実施します。
単純化のため、サンプルとインプットの両方のCtが同じ20サイクルだったとすると、%インプットは10%になります。
それぞれDNA量を計算すると、インプットサンプルは100 μlの出発材料を含むので、
= 100 μl ×(100 μg / ml)= 10 μg
ChIPサンプルについて、出発材料が1 mlですので、
= 1 ml ×(100 μg / ml)= 100 μg
よって、ChIPの後、サンプルとインプットが同じCt値(同じ量)なのであれば、100 μgの中から10 μgの目的タンパク質だけが濃縮されたことが示されたことになり、抗体によって10%が沈殿された(回収された)ことになります。
また、これにより入力サンプルのDNA量の違いによるバックグラウンドとChIP実験により生じるバイアスを正規化することが可能です。出発物質を用いているため、確実にCt値が得られ、偽陽性が出にくい長所があります。副産物的な効果として、qPCRに使用するプライマーの特異性の確認にもなります。
Ct値からChIP-qPCRの結果を計算する
さて、一般には、インプットは免疫沈降したサンプルと比べて濃度が濃いため、目的の免疫沈降サンプル量の1%のサンプルを取ってqPCRを行います。インプットとして取る量は、試料から得られるクロマチンDNAの量に応じて調整してください。
下にCt値からDNAの回収率を得るための計算方法を記しました。
1. 免疫沈降したDNAおよびインプットサンプルの、各プライマー対に対するqPCRの対数増幅期の閾値サイクル(Ct値)を記録します。
2. 以下の式を使用して、インプットDNAと比較した免疫沈降したDNAのコントロール領域の相対量(%回収率)を計算します:
%回収率= 2 ^((Ctインプット – 6.64) – Ct免疫沈降サンプル)×100
- Ct免疫沈降サンプルとCtインプットは、それぞれ免疫沈降サンプルDNAとインプットDNAのqPCRにおける対数増幅期からの閾値サイクルを示す。
- 2は増幅効率。
- 6.64はインプットの希釈率を補正するための補正係数。
注:この式は、PCRが100%効率的であると仮定しています(増幅効率= 2)。正確な結果を得るためには、所定のプライマー対による増幅効率は100%に近くなければならず、これは各サイクルにおいて生成物の量が2倍(E = 2)になることを意味しています。もし既知であれば実際の増幅効率を使用してください。
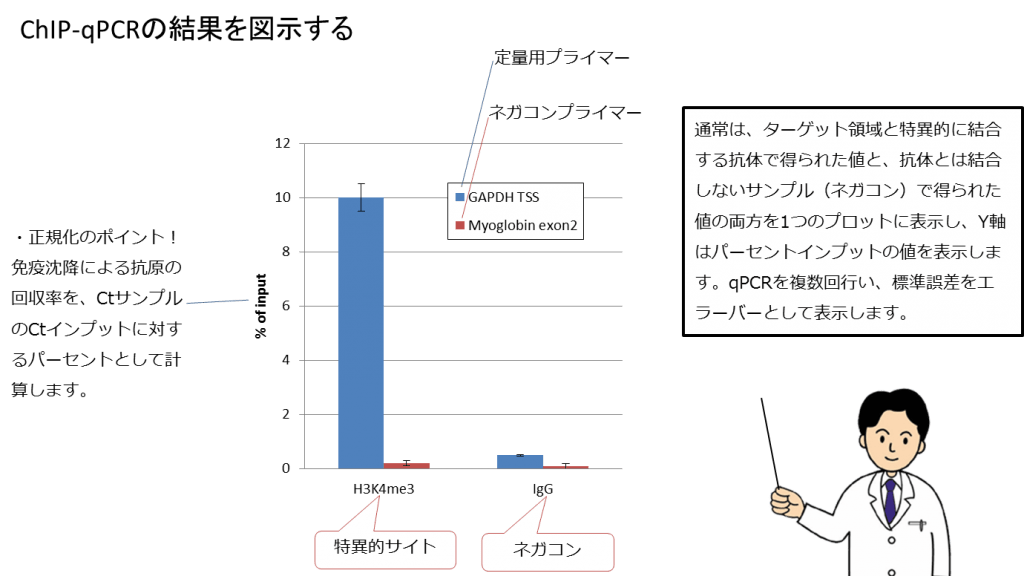
結果を作図します。
3. この公式は、インプットサンプル量がプロトコールに示されている通り免疫沈降サンプルの体積の1%であることが前提です(1μL インプットvs IPあたり100μLのクロマチン)。使用されるインプットの量が1%と異なる場合、以下のようにインプットの希釈(x)を補正するために、式に補正係数を導入する必要があります。
%回収率= 2 ^ [(Ctインプット-log2(X)) – Ct免疫沈降サンプル]×100
ここで、log2(X)はインプットサンプルの希釈倍率(倍)を表します。
例:
1免疫沈降あたり250μlのクロマチンを使用している場合、5μlのインプットサンプルは50倍希釈に相当します。補償係数はlog2(50)= 5.64に等しく、回収率を計算する式は次のようになります。
%回収率= 2 ^ [(Ctインプット-5.64) – Ct免疫沈降サンプル]×100
下のオンライン計算フォームにお手元の数値を入力していただきますと、%回収率が計算されます。
オンライン計算フォーム
測定数は最低2回以上、潜在的な外れ値を同定することができるようにするためには3重測定を実行することをお勧めします。可能であれば、結果はバックグラウンドシグナルと標準エラーと共に表示されることをお勧めします。
また、ネガティブコントロールとして、IgGを用いて免疫沈降したサンプルについても定量を行います。これは非特異的な目的DNA配列の沈降をコントロールするサンプルとなります。
iDeal ChIP-qPCRキットの特徴
DiagenodeのiDeal ChIP-qPCR kit(C01010180)(グローバルサイトにリンクします)は優れた特異性と感度を持つChIP-qPCR アッセイ用に最適化されています。高度に検証されたChIPグレード抗体と一緒に使用することで、優れた再現性のある結果を提供します。
iDeal ChIP-qPCR kitは、ネガティブコントロールとなるウサギIgGを添付しており、ご利用の抗体で生じる非特異的反応をモニターしていただけます。
繰り返しの実験で試薬が無くなってしまった場合は、ChIP-kit Customizer(グローバルサイトにリンクします)で無くなった試薬だけを補充することができ、大変経済的にご利用いただけます。