エピジェネティクス研究は、異なる転写パターン、遺伝子発現およびサイレンシングを引き起こすDNA上のメチル修飾や、 クロマチン の変化を対象としています。
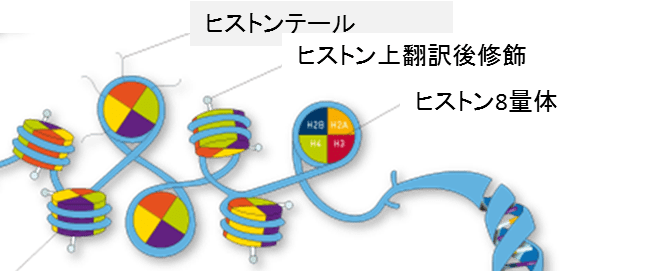
クロマチンはDNAおよびヒストン蛋白質によって構成されています。 各ヒストンコア蛋白質(H2A、H2B、H3およびH4)各2組8量体の高次構造が、DNAに包まれ、ヌクレオソームコアを形成しています。ヌクレオソームは、転写タンパク質のDNAへの接近やクロマチン再構成因子を制御します。
クロマチンを構成するヒストン上の翻訳後修飾の状態や近傍の転写因子の状態を、クロマチン免疫沈降によって確認することができます。
クロマチン免疫沈降(ChIP)は、関心対象の特定の蛋白質あるいは修飾に対するゲノム結合部位の位置を解明するために使用される方法であり、遺伝子発現の制御に関する非常に貴重な洞察を提供します。ChIPは特定の翻訳後修飾を含むクロマチン断片の選択的濃縮を行います。
ホルマリンでDNAとヒストンなどのDNA結合タンパク質を架橋した後、超音波処理によってDNAを断片化し、可溶性のクロマチンにします。これ をメチル化ヒストンやアセチル化ヒストンに対する抗体で免疫沈降します。免疫沈降物からDNAを精製し、次世代シーケンス(ChIP-seq)やリアルタイムqPCR(ChIP-qPCR)によって目的とするDNA領域がどれだけ濃縮されているかを解析します。
これにより、内在性のクロマチン領域にどれだけアセチル化されたヒストンや転写因子が結合しているかを調べることができます。ChIPは、このような利点から転写制御機構の解析において非常にポピュラーな方法となっています。
ChIPで明らかにできること
- 転写因子とヒストンのゲノムワイド結合部位を突き止める
- 特定のタンパク質に結合したDNA配列を明らかにする
- 疾患におけるDNA-タンパク質相互作用の理解
- 遺伝子発現・転写およびクロマチン構造に影響を与えるヒストン修飾の解明
エピゲノム研究において、クロマチン免疫沈降で得られる修飾情報はこれまでのエピゲノム情報に新たな次元を加えるもので、細胞間における発現の差異に対する新しい知見を与えるものです。
クロマチン免疫沈降を用いていろいろな状態の細胞の転写状況を調べることができるようになりましたが、必要な細胞数を集めるのが研究のハードルとなっています。
近年、この問題を解決するATAC-seq (Assay for Transposase-Accessible Chromatin with high throughput sequencing)と呼ばれる方法が開発され、これにより500−50000個の細胞があれば、細胞の特定の状態で活動可能になっている全遺伝子領域をマップすることが可能となってきました。
原理は、タンパク質の結合していない裸のDNA部位に選択的に挿入されるトランスポゾンを使っています。核を抽出・可溶化して、これにTn5 トランスポゼースと遺伝子シークエンスに用いるプライマー配列を挿入したトランスポゾンを混ぜて感染させます。
すると、染色体の裸のDNA部分にトランスポゾンが飛び込み、これによりゲノム全領域の中でオープンクロマチン領域だけを標識することができます。
この標識はシークエンスプライマーになっているので、この標識部位を次世代シーケンス(NGS)でシークエンスすることによって、転写が活性化されている場所とその頻度を調べることができます。
この手法では超音波処理によるDNAの断片化や抗体によるプルダウンが必要でなく、抗体のない種に対しても実験可能である反面、オープンクロマチンを網羅的にカバーする手法であるためターゲットを絞った解析には不向きという欠点もあります。
また、希少な断片化クロマチンにトランスポゼースとタグを入れたシーケンシングプライマーを混ぜて直接クロマチンからNGSライブラリを構築できるChIPmentationという派生技術も現れています。
必要な細胞数が少なく済み、にもかかわらず高い品質のデータが短い所要時間で得られるため、ガンなどの疾病に関わる希少な組織を用いた、遺伝子発現調節の研究に用いられています。
従来のキットにおいても、10000個の細胞があればChIP-seqが可能なキットが登場しており、さらなるエピゲノム研究の進展が期待されています。