5)ステップ5
定量PCR分析
注:各プライマーペアについて、免疫沈降したサンプルおよびネガティブIgGコントロールの横にインプットDNAの反応を行ってください。
IPしたDNAと対応するINPUT(ステップ4)のアリコートをとり、ChIP-seqグレードの水を用いてそれらを1/10に希釈します。
以下のようにqPCRミックスを調製します(付属のコントロールプライマーペアを使用して反応量20μlとする)。
・10倍量の2×SYBR®Green qPCRマスターミックス
・1μlのプライマー対
・4μlの水
・5μlの希釈された免疫沈降またはインプットDNA
次のPCRプログラムを使用してqPCRを実施します
注:これらの条件は、マスターミックスの種類、使用したqPCRシステム、およびユーザー提供のプライマーペアに応じて、最適化が必要な場合があります。
| ステップ | 時間・サイクル数 | 温度 | |
| 1. 変性 | 3-10分* | 95℃ | |
| 2. 増幅 | 30秒 | 40サイクル | 95℃ |
| 30秒 | 60℃ | ||
| 30秒 | 72℃(蛍光データ取得) | ||
| 3. 融解曲線** | qPCRマシンの製造元の推奨条件に従ってください | ||
* ホットスタート用Taqポリメラーゼの活性化時間に関するサプライヤーの推奨事項をご確認ください
**プライマー対が単一の特定の産物のみを増幅することを確実にするために、qPCRマシンの製造元が推奨するプロトコルに基づいて融解曲線の確認、検査を実施してください
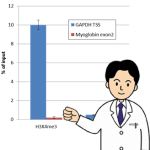
免疫沈降したDNAおよびインプットサンプルの、各プライマー対に対するqPCRの対数増幅期の閾値サイクル(Ct値)を記録します
インプットDNAと比較した免疫沈降したDNAのコントロール領域の相対量(%回収率)を計算します
ポジティブコントロールの結果について、バックグラウンドが低く、期待される濃縮が見られた場合は、適切なキットやプロトコル(iDeal ChIP-seq for Histonesには含まれていません)を使用してライブラリ調製に進んでください。
iDeal ChIP-seq for Histonesの自動化および手動プロトコルは、Illumina® HiSeq Next-GenシーケンサーにおけるChIP-seqにおいて検証済みです。
Illumina®シーケンサーと互換性のあるライブラリ作製のために、MicroPlex Library Preparationキットv2(カタログ番号C05010012、12個のインデックス、またはC05010014、48個のインデックス)またはiDeal Library Preparationキットx24(インデックスプライマーセット1を含む) (カタログ番号C05010020)などのDiagenode製キットを使用されることを推奨します。