ChIPアッセイの実験手順はいくつかのステップに分かれており、それぞれに係るパラメーターが多いため、ポイントを押さえないとなかなか一度で実験を成功させることは困難です。
そこでここでは、それぞれのステップに分けて実験のポイントを示し、押さえておくべき基本の注意事項を示しました。
実験を進めるうえでうまくいかない場合にも、このチャプターを見直すことによってトラブルシューティングにつなげていただければと思います。
Step 1. DNA-タンパク質のクロスリンク
クロスリンクによる固定化は、可逆的なDNA-タンパク質結合を形成するホルムアルデヒドを用いることによって行われます。
ホルムアルデヒドは細胞膜を迅速に透過し、インタクトな細胞内の密接に関連したタンパク質同士をクロスリンクします。ホルムアルデヒドによるクロスリンクは、直接相互作用する2分子の分析には理想的な手法です。
しかし、さらに高次な、あるいは動的な相互作用にかかわるタンパク分子については、革新的な二重クロスリンクChIP固定化試薬であるChIP Cross-link Gold (Diagenode C01019021)(グローバルサイトにリンクします)のような効率的なタンパク質-タンパク質安定化能を有する他の固定化試薬を検討すべきでしょう。
あるエピジェネティックマークが、他のエピジェネティックマークと比較してタンパクの結合部位の特定がしにくいケースも存在します。
相互作用の弱い、またはまれなタンパク質-DNA結合を研究する場合に、迅速にクロスリンクするため固定化試薬を直接メディウムに加えて行うことも考慮すべきでしょう。
アセチル化ヒストンのマークを研究する場合、細胞はクロスリンク前にトリプシン処理を行い懸濁状態にしておきます。
一般的に、標準的なホルムアルデヒドを用いた単一ステップでのクロスリンク化プロトコールを用いた場合、転写因子(10〜20分)よりもヒストンマーク(8〜10分)の方が短い時間でのクロスリンクが必要です。
新鮮な分子生物学用ホルムアルデヒドの使用は非常に重要です。これに対し、メタノールフリーの試薬を使用することは必須ではありません。毎月ホルムアルデヒドのストックを新しく調製してください。これにより、ChIP実験の高いアッセイ間再現性が確保されます。
常に細胞株のクロスリンク時間のタイムコースを取ってください。使用する細胞株と目的のエピトープの最適なクロスリンク時間を経験的に決定するためです。細胞株およびエピトープは、それぞれクロスリンク効率およびクロスリンクに対する感受性が大きく異なります。
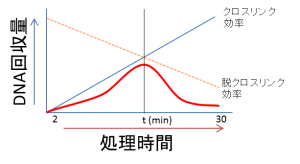
図:クロスリンク処理時間とDNA回収量(模式図)
クロスリンク時間と温度を正確に実施してください。ホルムアルデヒドによるクロスリンクは、時間および温度に依存するプロセスです。 より高い温度およびより長い時間でより強いクロスリンクが達成されます。RTまたは37℃、5または15分(ターゲット分子および細胞タイプに応じて変更する)を使用する場合でも、常に同じ温度と時間が維持されていることを確認してください。
| ターゲット 固定化試薬 | ホルムアルデヒド | ChIP Cross-link Gold C01019021 (グローバルサイトにリンクします) |
| ヒストン | ○(8-10分処理) | × |
| DNAに直接結合した転写因子 | ○(10-20分処理) | × |
| 間接的高次結合・動的結合 | ○(10-15分処理) | ○(30-45分処理) |
細胞固定化・クロスリンクプロトコール
ダイアジェノードではChIPにおける細胞固定化・クロスリンクに際しては、分析対象に応じて推奨プロトコールをご用意しております。以下のリンクよりダウンロード可能ですので是非ご覧ください。
Step 2. 細胞溶解とクロマチン断片化
溶解のステップでは細胞膜を界面活性剤ベースの溶液で可溶化することによって細胞成分を遊離させます。それにより、クロスリンクしたタンパク質-DNA複合体を細胞または組織から溶液中に抽出します。
細胞溶解を完了するためには超音波処理が必要です。一般に、クロスリンクの条件が強くなればなるほど、細胞溶解および超音波処理に使用すべき条件が厳しくなります。
細胞の溶解方法として、SDS含有緩衝液を用いて1段階溶解を行うことが可能です。1段階の溶解は、限られた数の細胞(1×106個未満)で開始する場合に最も適切です。
また、最初に細胞膜を、非イオン性界面活性剤を含む緩衝液を用いて溶解した後、単離した核をSDS含有緩衝液で処理する2段階溶解で溶解することも可能です。 2段階溶解は、可溶性の細胞質に存在するタンパク質の大部分を除去できます。これにより、超音波処理効率を改善し、ChIPアッセイのバックグラウンドを減少させ、感度を増加させることが可能です。
細胞数が多い場合(1×106個以上)や、あるいは強い固定化が行われた細胞や分析が困難な細胞では、核分離による2段階の溶解が適切です。
Step 3. 免疫沈降法
免疫沈降法(IP)とは、磁性ビーズなどに固定化された特異的抗体を用いた、小スケールの抗原タンパク質のアフィニティー精製を指します。
断片化クロマチン溶液から目的のヒストンマークを持つクロマチンや転写因子を回収するため、特定のタンパク質あるいはヒストンマークに対する抗体を断片化クロマチン溶液に加え、抗原抗体の複合体を形成させます。同時に複合体を抗体に強い親和性を持つprotein Aやprotein Gを固定化したビーズを用いて回収します。
その後、固定化された抗体に抗原が結合した複合体を洗浄した後、ビーズを溶液中から回収します。
DiaMagプロテインAコーティング処理済磁気ビーズ(グローバルサイトにリンクします)は、ウサギポリクローナル抗体、マウスIgG2a、IgG2bおよびIgA、モルモットIgG、イヌIgG、ブタIgGの免疫沈降に適しています。
クロマチン免疫沈降、特にChIP-seqで使用される抗体の品質は実験の成功に不可欠です。標的タンパクを特異的に認識する確認済みの抗体のみを使用することを推奨します。Diagenodeは、ChIP-seqで特異性が確認されており、幅広く検証された高性能抗体を提供します。各バッチは検証され、バッチ固有のデータはウェブサイトwww.diagenode.comで入手できます。
Step 4. 溶出、脱クロスリンクおよびDNA精製
抗体との複合体として回収されたクロマチンからDNAを精製する必要がありますが、クロスリンク法では、DNA-タンパク質間のクロスリンクを解消させる必要があります。
ホルムアルデヒドによるクロスリンクは、SDSの存在下での加熱による加水分解で分解されます。
この時、RNA分解酵素やタンパク質分解酵素を同時に作用させ、解離したDNAだけDNA精製用ビーズを用いて精製を行います。
ChIPの後、Thermo Fisher ScientificのQubit®システム・dsDNA HS Assay Kitなどの高感度メソッドで免疫沈降したDNAの濃度を測定してください。
PicoGreen®も適していますが、NanoDropのようなUV分光光度法は、通常、十分な感度を有していません。
ほとんどの場合、定量には免疫沈降したサンプルの約10%を使用すれば十分です。
DNA収量は、細胞の種類、使用される抗体の品質およびエピトープの種類などの異なる因子に依存します。例えば1×106個のHeLa細胞に対し陽性対照H3K4me3抗体を用いて得られる予想DNA収量は約10 ngです。
Step 5. 定量PCR分析、NGS分析
シークエンシングの前に、少なくとも1つのポジティブコントロール領域と1つのネガティブコントロール領域を使用し、SYBR®Green qPCRでインプットおよび免疫沈降したサンプルを分析して濃縮度を決定することを推奨します。
本キットには、キットで提供されるコントロール抗体(H3K4me3 ChIP-seqグレード抗体)の結合領域に対する、陽性(GAPDH TSS制御領域)および陰性(Myoglobin Exon 2)の2つの領域を標的とする2つのプライマーペアが含まれています。試験対象となる特異的抗体に対しては、特異的なコントロールプライマーをユーザーが設計する必要があります。各プライマーペアについて、免疫沈降した試料の横にインプットDNAを泳動します。配列決定のために十分なDNAを残すために、qPCRには免疫沈降された全DNAの10%以上を使用しないことを推奨します。
PCR反応を十分な回数実行するためにDNAを(1/10またはそれ以上)希釈することができます。最低2回はPCR反応を行われるべきですが、潜在的な外れ値を同定することができるようにするためには3重測定を行うことが推奨されます。
製品案内
ChIP Kit Customizer
キットに含まれる、最適化された試薬はもし実験で切れてしまった際にまた個別に注文していただけます。ChIP Kit Customizerで、実験に必要な最適化済みの試薬をサイト上で手軽に注文し、無駄なく手に入れることが可能です。ログインによってフルに機能をご利用できます。