RNA-seqと発現解析
次世代シーケンシングによるDNA解析が身近なものになった現在、RNA-seqは網羅的遺伝子発現解析の手法として様々な生物種を対象に行われています。
RNA-seqは試料から抽出したmRNAを逆転写して得られたcDNAをライブラリー化し、次世代シーケンサーで解析することによって発現遺伝子の特異的配列と頻度を網羅的に取得する手法です。しかしながら、統計的に有意な比較解析データを得るためには多くのサンプルの定量データを取得する必要があるため、解析コストの低減が課題として挙がっていました。
ハイスループット3'mRNA-seqとは
ハイスループット3’ mRNA-seqは、多数のサンプルを用いた比較遺伝子発現分析を経済的に実現するためのソリューションです。本法はEPFL(スイス・ローザンヌ)のBart Deplancke教授によって開発されたBRB-seqプロトコールを利用しており、大規模な調査研究で信頼性の高い遺伝子発現定量を可能にします。
特に、対象となるサンプルについて複数の条件を評価する場合や、大規模な生物学的レプリケートセットを導入した発現比較分析の統計的検出力を向上させるのに最適です。
BRB-seqテクノロジー
RNA-seqでは、ダイレクショナルdUTP法(TruSeq法)がゴールドスタンダードとしてバルクトランスクリプトーム解析に広く用いられていますが、サンプル一つ一つに対し処理を行う必要があり、労力がかかります。また、mRNA全長を読むため、解読塩基数が多くなりコストが上昇することも課題でした。
ハイスループット3’mRNA-seqで導入されたBRB-seq(Bulk RNA Barcoding and sequencing)は、mRNAの計数に特化しており、mRNAの3’側配列のみ含まれるcDNAライブラリーを構築します。
最大の特徴は、個々のサンプルに対し、逆転写の段階で分子バーコードを導入する初期マルチプレックス化によって、後のステップを一括処理することが可能となる点です。これにより多サンプルの処理労力が軽減されるだけでなく、ライブラリー調製とシーケンシングコストが大幅に低減されます。
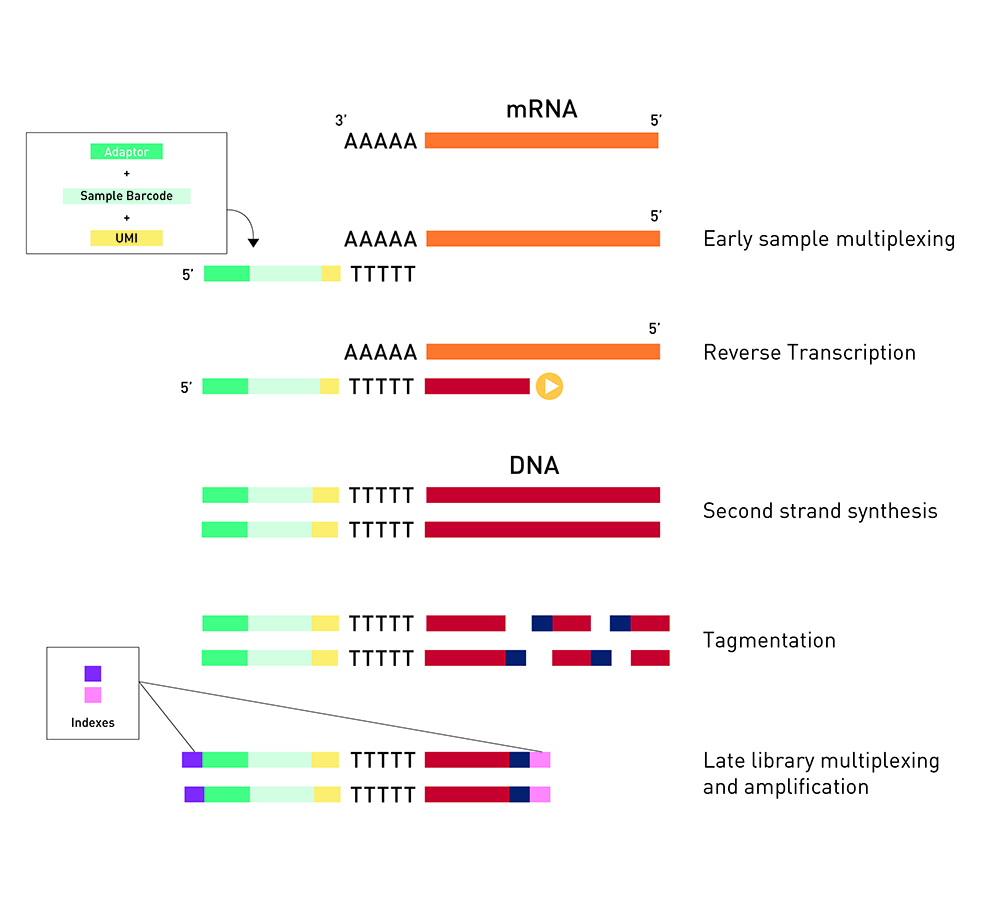
分子バーコードについてはサンプル識別用のバーコードと共に、本法に最適化されたUMI (Unique Molecular Index)が導入されており、データからPCRバイアスを除去することが可能です。
本法のように3’側を濃縮してシーケンスすることにより、必要な解読塩基数も低減させることが可能です。解読された塩基のうち、得られた配列の75%が遺伝子上にマップされ、従来のRNA-seqの手法と同じシーケンス深度で同等の遺伝子検出力を示します。また、従来法と発現変動遺伝子を比較したところ高い一致率を示し、確度の高い発現変動の検出力を有することが示されました。
労力とコストが軽減され、従来の手法より処理可能なサンプル数が格段に向上したハイスループット3’mRNA-seqは、解析可能な発現変動遺伝子数においてマイクロアレイを代替でき、遺伝子発現プロファイリング・発現変動解析において良好なパフォーマンスを提供できる手法となります。