Next Generation Sequencer(NGS, 次世代シーケンサー)とは
次世代シーケンサー(NGS, Next Generation Sequencer)は、ランダムに切断された数千万―数億のDNA断片の塩基配列を同時並行的に決定することができる装置です。
断片化した1本鎖のDNAに対して、断片の相補鎖を合成しながら決定する手法(SBS, Sequence-by-Synthesis法)によって、塩基配列を決定します。
当初はゲノム配列のハイスループットな解読を目的に作成された手法ですが、読み取り深度を深めることによって頻度の少ない変異や微量DNAの検出や、ChIPアッセイで得られたDNA断片の網羅的配列決定・解析、転写産物の網羅的配列決定・解析など、広く使われています。
現在、ここに紹介したSBS法を使用したHiseqやMiseqなどのシーケンサーを発売しているイルミナ社、Ion Torrentシリーズを販売しているサーモフィッシャーサイエンティフィック社の製品が多くの研究室で使用されています。また、Pacific Bioscience社が販売するロングリードシーケンサーであるPacBioもその1リード当たりの決定塩基数の多さより注目されています。
ChIP-seqとは
クロマチン免疫沈降(ChIP)法とハイスループットの大規模並列シーケンシング(次世代シーケンシング、Next Generation Sequencing)を組み合わせた検出法ChIP-seqアッセイは、エピゲノム研究、すなわちゲノムワイドでのタンパク質-DNA相互作用のマッピング研究のための主要な方法の1つになっています。
目的のタンパク質との相互作用により特異的抗体で免疫沈降したクロマチンDNAを網羅的にシーケンシングすることによって、細胞の特定の状態においてゲノム上での目的のタンパク質とDNAとの相互作用部位を明らかにすることが可能です。
ChIP-seqからは重要な情報が得られる反面、十分な量のDNAライブラリ調製のため、大量の細胞や組織が必要で腫瘍や胚組織などには適していない欠点があります。
一般には少量の細胞・組織からでもクロマチンDNAを精製でき、シーケンス用ライブラリー作製のためのアダプターを接続可能なキットを用いてサンプルの調製を行います。
ChIP-seqワークフロー
クロマチンDNAの抽出・精製
初めに、目的の細胞を可溶化し、超音波破砕機・Picoruptorなどを用いてクロマチンDNAを断片化後、ChIPキットなどを用い、免疫沈降(クロマチン免疫沈降)を行って濃縮、精製します。
NGSライブラリー調製
次に精製したクロマチンDNAからNGSライブラリーを作製します。
主要な次世代シーケンシングプラットフォームのほとんどはシーケンシングの前に、特異的なアダプターオリゴを断片化したDNAまたはRNA にライゲーションする必要があります。
断片化されたクロマチンDNAはそれぞれ末端修復(エンドリペア)され、平滑末端化されます。
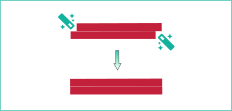
次のステップとして、平滑化リン酸化DNAフラグメントの3 ‘末端にdAMPを加えてコンカテマー化を防止し、相補的なdTオーバーハングを有するアダプターのライゲーションを可能にするA-テーリングを行います。
A-テーリングを行ったDNAにアダプターをライゲーションします。
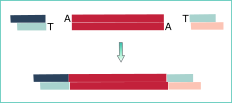
最後にDNAをクラスター化するためにPCR増幅を行います。
増幅前または増幅中に、シーケンスのマルチプレックス化を図るため、バーコードを組み込むこともあります。


これらの処理を行うため、初発DNA量は多く、DNAのロスはできる限り少なく抑えることが望まれます。
多くの場合はライブラリー調製キットが存在しています。DiagenodeのMicroPlex Library Preparation kit(グローバルサイトにリンクします)は手順を少なくすることによりDNAのロスを最小限にでき、ChIPのように少ない初発DNA量からスタートする時のNGSライブラリー調製に最適です。
また、希少な断片化クロマチンにTn5 transposaseとタグを入れたシーケンシングプライマーを混ぜて直接クロマチンDNAからNGSライブラリーを調製できるChIPmentation(グローバルサイトにリンクします)という技術も現れています。
次世代シーケンシングとデータの解析
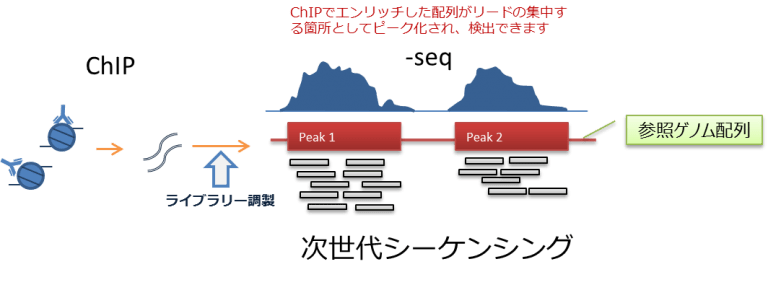
調製したサンプルを、次世代シーケンサーを用いて同時並行・網羅的に配列決定を行い、得られたデータを分析します。
読まれた配列をレファレンスゲノム配列上にマッピングすることによって、転写因子の結合部位を同定し、結合モチーフを発見することが可能です。
ATAC-seqとは
近年、ATAC-seq (Assay for Transposase-Accessible Chromatin with high throughput sequencing)と呼ばれる方法が開発され、これにより500−50000個の細胞があれば、細胞の特定の状態で活動可能になっている全遺伝子領域をマップすることが可能となってきました。
原理は、クロマチンの結合していない裸のDNA部位に選択的に挿入されるトランスポゾンを使っています。核を抽出・可溶化して、これにTn5 transposaseと次世代シーケンスに用いるプライマー配列を挿入したトランスポゾンを混ぜて感染させます。
すると、染色体の裸のDNA部分にトランスポゾンが飛び込み、これによって、ゲノム全領域の中でプロモーター、エンハンサー、およびインシュレーターなどが結合する遺伝性制御にかかわるオープンクロマチン領域だけを標識することができます。
この標識はシーケンスプライマーになっているので、この標識部位を次世代シーケンス(NGS)でシーケンスすることによって、ゲノムの中で転写が活性化されている場所とその頻度を調べることができます。
たとえばATAC-seqで推測されたオープンクロマチン領域に結合する転写因子について、転写因子特異的な抗体を用いてChIP-seqで解析することもできます。
Diagenodeでは、サンプル調製の専門知識と経験を生かし、受託ChIP-seqプロファイリングサービス、受託ATAC-seqサービスをお客様に提供しております。どうぞご利用ください。
製品案内
Diagenodeキットセレクタ
Diagenodeグローバルサイトでお客様のニーズに合わせたキットの選択をしていただけます。リンク先で用途をクリックし、詳細をご覧ください。

True MicroChIP kit
少ない細胞数からのChIPアッセイを可能とするDiagenode社製ChIPキットです。本キットを使用したChIP-seqライブラリ構築の手順を動画で見ていただけます。

iDeal ChIP-seq kit for Histones
Diagenodeの検証済みiDeal ChIP-seqキットは必要な試薬全てを含んでおり、再現性高く結果を得ることができます。複数のヒストンマークについて確認済みのキットです。

iDeal ChIP-seq kit for Transcription Factors
DiagenodeのiDeal ChIP-seq for Transcription Factorsキットは、安定した転写因子ChIP-seqの結果を実現するために特化されたキットです。実験結果の例とお客様の声をご覧ください。

MicroPlex Library Preparation Kit v2 (12 indices)
MicroPlex Library Preparation™キットは、ChIP-seqで検証され、ピコグラムオーダーのDNAからライブラリーを作成できる唯一のキットです。製品の紹介と製品に関する研究者の声をご覧いただけます。