ライブラリー調製 (15)
【FAQ】クロマチンのせん断サイズは500bpでよい?
ChIP、およびライブラリー作製のワークフローはより小さな断片が濃縮されるようなフローとなっております。
これに加え、Illuminaのフローセル上ではライブラリー断片が小さいほうがクラスター形成の効率が高いです。
これらの効果によって大きなサイズのクロマチン分画とのバランスを保っているので、十分良好なカバレッジと分解能が得られます。
【FAQ】CUT&Tagのオフターゲット効果
pA-Tn5トランスポザーゼ融合タンパク質は、目的の分子を認識する抗体に結合し、適切な洗浄ステップ後、Mg++イオンの添加後にタグメンテーション活性の活性化が起こるので、オフターゲットは最小限であることが期待されます。
【FAQ】Tn5 transposaseの切断部位
切断部位のタンパク質からの距離について明確な値は知られていませんが、アダプター配列およびインデックス配列を含めて150~2000bpの範囲内のライブラリーが通常得られています。
Tn5トランスポザーゼ作用後のほとんどのDNA断片長は約500bpですが、これはゲノムのエピジェネティックマークやサンプルの種類によって異なる場合があります。
【FAQ】CUT&Tagで配列バイアスはかかる?
CUT&Tagライブラリー作製では、目的の標的に対する一次抗体の結合部位に連関する配列がターゲットで、Tn5による断片化部位の配列は比較的重要ではないため、バイアスは無視できます。
ChIPと比較して、ヒストンマーク特異的な結合の検出感度が高くなるので、ChIPよりも少ない細胞を使って実験することができます。
【FAQ】植物サンプルのタグメンテーション
残念なことに、植物から核を単離するための検証済みのLysis試薬は弊社では有しておりません。
弊社のATAC-seqキット附属の核単離試薬を用いて植物のATAC-seqを行い、非常に良好な結果を確認しておりますが、正確な分離プロトコールを植物試料タイプごとに確立する必要があると考えられます。
【FAQ】ライブラリー定量にBioanalyzer?
KAPA Library Quantification kitを用いた定量法をご使用ください。
KAPA Library Quantificationはタグが付けられ、かつアダプターを有する断片のみを増幅し、濃度を正確に算出できます。
バイオアナライザーだけでは、サンプルの量を正確に反映できない場合があります。
【FAQ】ライブラリー定量にQubit?
Qubitで算出されるDNA量の単位は、ng/μLの濃度になるため、断片サイズを測定する方法を加えないと、ライブラリーによく使われるnMとして使用する上での理由づけが難しい場合があります。
もうひとつの潜在的な問題は、検出原理的にアダプターダイマーが定量サンプル中に検出されうることであり、これがQubitの結果に偏りを与える可能性があります。
【FAQ】CUT&TagでATACシグナルが混入することはある?
ATAC-Seqプロファイルは一般にヌクレオソームのラダーのように見えるため、CUT&Tag実験では適切な陽性コントロールを用いて識別できるようにすることが有用です。
ポジティブコントロールとしては、シグナルがATAC-seqシグナルと被りにくいH3K27me3が特に適しています。H3K4はATAC-seqシグナルに類似しているため、避けたほうが良いでしょう。
また、CUT&Tagでは、pA-Tn5活性を厳密に制御することによって、ATAC-seqシグナルが問題となることを回避できます。
洗浄によって非結合型の酵素を反応溶液から除去した上で、Mg2+を添加して抗体結合型pA-Tn5の活性化を行うことが可能なため、ATAC-seqシグナルを最小化することが可能です。
また、ATAC-seq Signalと反相関するH3K27me3陽性対照(および対照プライマー)をコントロール実験として含めることで、ATAC由来の異常なシグナルを検出することが可能です。
【FAQ】DNA精製キットの違いについて
DiagenodeのIPure kitは、磁気ビーズを使用したDNA精製キットであり、ChIP、MedIP、およびCUT&TagからDNAを抽出するために特別に最適化されています。
磁気ビーズを使用すると、DNAのみを選択的に分離できるため、DNA精製の再現性が向上します。
このシンプルなプロトコルは、あらゆるダウンストリームアプリケーション(次世代シーケンシングなど)に対応できる精製度の高いDNAを提供します。
フェノール-クロロホルム抽出と比較すると、IPureテクノロジーには、毒性がなく、複数のサンプルで実行するのがはるかに簡単であるという利点があります。
DNAは>25μlで溶出できますので、Ideal ChIP-seq kitなどに最も適切に使用していただけます。
DiagenodeのMicroChIP DiaPureカラムは、非常に少量のDNAの精製と溶出に最適化されています。
プロトコールは、低インプットChIP(True MicroChIPキットを使用するなど)やCUT&Tag(DiagenodeのpA-Tn5を使用するアプリケーション)などの様々なエピジェネティクス実験アプリケーションで検証されていますが、他の多くのアプリケーションとも互換性があります。
DNAは、6μlまでの高濃度で溶出でき、あらゆる下流アプリケーション(NGSなど)に適しています。
【FAQ】ChIPmentationによるライブラリー調製について
ChIPmentationのコア技術であるTagmentationによるライブラリー調製はライゲーションベースのライブラリー調製と比べて早く、また少ない手順で進めることが可能です。したがって大きいコホートやサンプル当たり沢山のChIPターゲットが存在する場合に非常に有用です。
【FAQ】Microplexライブラリー調製キット
MicroPlexライブラリー調製キットはdsDNA 50pgから50ngの範囲で、イルミナNGSプラットフォーム用のシーケンス対応ライブラリーを調製可能です。
高速でシンプルな3ステッププロトコルを使用し、簡便にライブラリーを調製していただくことが可能です。
【FAQ】ChIPmentationによるChIPの自動化について
はい、ChIPmentationの最初の超音波処理による断片化ステップはChIPの場合とまったく同じですので、クロスリンクとソニケーションを行ってください。その後の免疫沈降ステップがライブラリー調製のステップと統合化されています。
また、ChIPmentationステップはSX-8Gのプログラムに完全に組み込まれています。SX-8Gを使って自動化されたChIPmentationキットの実験を行っていただけます。
【FAQ】植物サンプルとMicroplexキットについて
Microplex Library Preparation kit v2は50 pgの全ての種のChIP済みDNAから次世代シーケンス用ライブラリーを調製していただけるキットです。
DiagenodeではMicroplexキットに互換性のあるChIP-seq用ChIPキットを取り扱っております。
サンプルの種類とターゲットの種類(ヒストン/転写因子)に応じて、下記キットを使用することにより、Microplexキットに使用できるクロマチンDNAを調製していただけます。
- iDeal ChIP-seq Kit for Transcription Factors (Cat. No. C01010055)
- iDeal ChIP-seq Kit for Histones (Cat. No.C01010051)
- True MicroChIP kit (Cat. No. C01010130)
- Universal Plant ChIP-seq Kit (Cat. No. C01010152)
(これらのキットはマニュアルでの実験および自動ロボットによる実験に対応しています。)
従って、植物サンプル由来のサンプルには、Microplexキットに互換性のあるUniversal Plant ChIP-seq Kitをご使用ください。
【FAQ】サンプル量とNGSライブラリー調製について
少なくとも同じの量のクロマチンを用いてChIPを始め、得られたDNAの濃度をさらに揃えることをお勧めします。
実質的に異なる量でChIPを開始する場合、ChIP時に免疫沈降の効率や特異性がサンプル量に影響を受け、最終的に無視できないバイアスを結果に生じさせる場合があるためです。
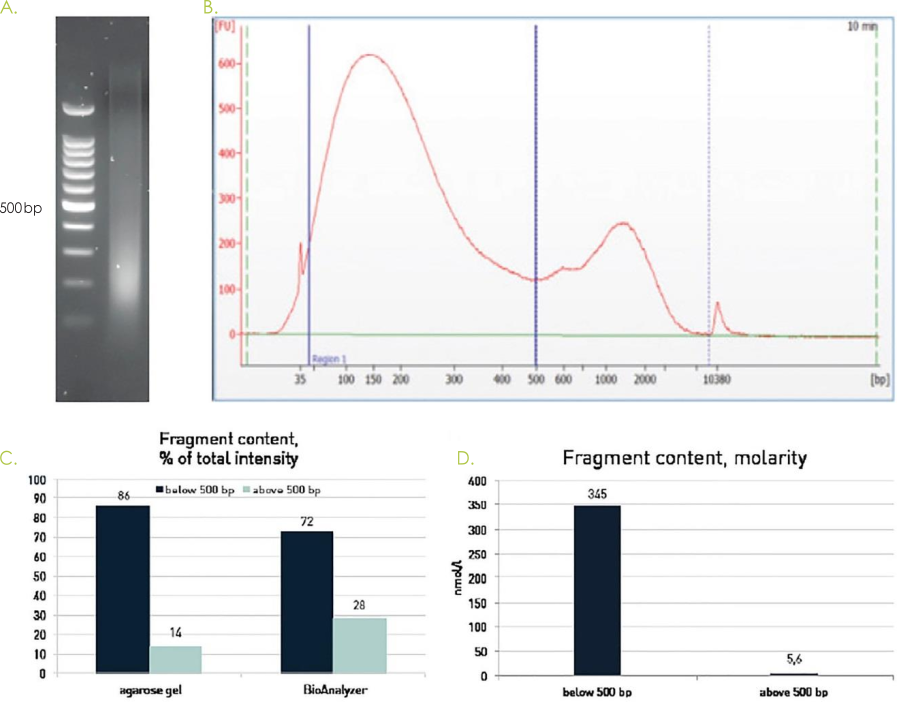
【FAQ】Bioanalyzerでのピークの出方について
BioAnalyzerでサンプルを検定するとしばしば高分子側に2番目のピークが現れます。
この2番目のピークは通常対数スケール表示に伴うピークで、500 bp以上のX軸のデータが圧縮表示されることによって、高分子側の低くなだらかなピークが視覚的に大きなピークとして表示されます。
また、低分子量の分子と異なり、高分子量の分子のほうがシグナルを強く発するため、高分子側のピークが強調される傾向もあります。
BioAnalyzerのソフトウェアはピーク面積を分子数で表示する機能を有しており、これを使用すれば2番目のピークは通常かなり改善されます。
よって、2番目のピークは通常ライブラリー調製に影響は与えません。
また、ライブラリー調製時には低分子側の断片のほうが効率良く取り込まれるため、高分子の断片は調製中に大部分が取り込まれずに除去されます。
アガロースゲル電気泳動ではこのような断片長、およびSDS・タンパク質・共沈剤などの不純物に由来する誤検出が少ないため、DiagenodeではクロマチンDNA断片のサイズ確認にはアガロースゲル電気泳動の使用を推奨しております。
Figure 5. アガロースゲルおよびBioAnalyzerを用いた断片化クロマチンDNAのサイズ評価。
HeLa細胞をホルムアルデヒドで固定し、染色体をDiagenodeのプロトコルに従って調製した。 サンプルを、Picoruptorの30サイクルON / 30秒OFFで1.5 ml Picoruptor Microtubes with Caps(C30010016)を用いて超音波処理し、アガロースゲル(図A)、またはBioAnanlyzer、High Sens Agilent DNAキット(図B)によって測定した。 500bp以下および500bp超のフラグメント含有量を全表面積のパーセンテージとして算出した(図C)。図Dはモル濃度として計算されたフラグメント含有量を示す(BioAnalyzerトレースのみ)。