ChIP-qPCR (11)
【FAQ】ChIPの出発細胞数はゲノムサイズに応じて変える?
通常、細胞数をゲノムサイズに応じて増減する必要はありません。ほとんどの真核生物種では同じサンプル量で処理できます。もちろん、酵母や、細菌のDNAに結合したタンパク質にChIPを適用する場合は細胞数を増やすことも可能です。
植物の場合は、必要な組織の量は必ずしもゲノムサイズによらず、むしろ核/クロマチンの全体的な量によって異なるため、サンプルに応じて出発物質量は50mgから2gまでさまざまであることが多いです。Diagenodeでは植物の多様性に対応し使用できる作業フローを確立し、キット化していますので、これらキットもお試しください。
Universal Plant ChIP-seq kit (https://www.diagenode.com/en/p/universal-plant-chip-seq-kit-x24-24-rxns)
EasyShear Kit for Plant (https://www.diagenode.com/en/p/chromatin-shearing-plant-chip-seq-kit)
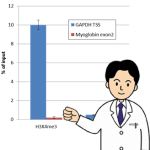
【FAQ】ChIP-qPCRでCt値が得られないサンプルの場合
一つの方法としては、「測定限界外」のCt値を仮に「>40」として値に40を取ることです。
すなわち、Ct=40をPCRサイクルで増幅が得られないサンプルとして定義します。
この値に基づき、サンプルの測定値を比較Ct法などを用いて相対定量計算を行います。
ここから言えるのは、サンプル測定値 vs. IgGコントロールによる正規化は通常ChIP-qPCRでは必要ないということです。
ChIP-qPCRでは免疫沈降サンプルのCtはインプットによって正規化されるのがより実用的です。
IgGのqPCRシグナルは通常免疫沈降シグナルよりかなり低く、全く検出されない場合もあるため、Ctを用いた正規化の計算には不向きと言えます。
特異性の評価については、別にIgGをインプットによって正規化したデータを使用して示すのが適しています。
【FAQ】ChIP-qPCR用プライマーペアの設計
プライマーを設計するときは、ターゲットの生物学的意味について推定することが避けられません。
最初に確認するのは、同じターゲットを測定したデータセットがすでに公開されているかどうかです。
まだ類似の研究がされていない場合には、ターゲットの生物学的機能から可能な増幅ターゲットが何であるかをある程度推測するか、プライマーアレイ(設計済みあるいはカスタムのプライマーペアセット)を使用する必要があるかもしれません。
ネガティブコントロールを設計する場合、通常、遺伝子間領域(intergenic regions, “gene desert” )をターゲットにすることができます。
併せて下のページも是非ご参照ください。
https://www.diagenode.com/jp/categories/primer-pairs(グローバルページにリンクします)
【FAQ】ChIP-qPCRとChIP-seqのデータの正規化
特に数量的データの正規化にはインプットからのリカバリー%をご利用ください。
IgGはバックグラウンドノイズがどの程度あるかについての指標にはなりますが、正規化に使用するにはインプットほど確度が高くありません。
qPCRの際は特にインプットを使用して正規化を行うことが重要です。
下のページについても併せてご覧ください。
【FAQ】ChIP-qPCRで使用するプライマーの選択
ChIPで使用するプライマーはゲノムDNA配列上のターゲットの結合する領域に対応した配列を使用する必要があります。mRNAをターゲットとするプライマーは通常ChIPには適していません。
良いChIP-qPCR用のプライマーをデザインすることはいくつかの理由から難易度が高いことが知られています。
以下の点にご注意ください。
・ChIP用のプライマーは限られた領域を検出する必要があるため、選択肢が少なく設計上の自由度が低い傾向にあります。特に転写因子のように結合領域が狭い場合、ある抗体では動作するプライマーが別のChIP実験ではうまく動作しない場合があります。
・qRT-PCRでは特異性を向上させるためにイントロンをまたぐプライマーがしばしば使用されますが、ChIPではゲノムDNAが鋳型なため、そのようなプライマーは使用できません。
・断片化クロマチンのサイズに合わせ、また非特異反応を上回る増幅を得るため、アンプリコンは短く(50-150 bp)設計することが求められます。
【FAQ】ChIP-qPCRのネガティブコントロールプライマー
シーケンスに進む前にChIPがうまくいっているかを確認する方法として、予想されるChIP濃縮産物のDNA上領域に設計したプライマー(ポジコン)と免疫沈降で濃縮されない画分のDNA上に設計したプライマー(ネガコン)を用いてqPCRを行い、濃縮効果を確認することができます。この方法はヒストンマークのほうがどの領域が濃縮されているかがはっきりわかるため、転写因子よりはヒストンマークの検出に適しています。
ネガコン領域は通常遺伝子間領域(intergenic regions, “gene desert”)から選定します。
コントロール領域選定の一例をあげると、H3K4me3修飾特異的なヒストンをChIPして得られたChIP DNAを検出する場合、最も発現している遺伝子(GAPDHのようなハウスキーピング遺伝子等)の転写開始部位特異的なプライマーをポジコン、目的の転写部位から離れた(5-10Kbp以上)部位に特異的なプライマーをネガコンとしてプライマーを設計してください。
得られた定量結果の正規化のため、インプットサンプルのqPCRも同時に実施することを推奨いたします。
【FAQ】ChIP-qPCRネガコンの設計部位
ネガコン領域はChIPで検出対象とするヒストンマークの修飾の種類に応じて選択します。
検出したいヒストンマークと相関しない(非活性、あるいはH3K4me3の場合転写が生じていない)領域を選択してください。
目的の部位から離れていてもタンパク質との相互作用が存在しうるため、部位をいくつか選択しテストしてください。
ネガコン領域として使用できる部位の一例をあげると、特異性細胞でしか発現しない遺伝子の転写開始部位、あるいは遺伝子間領域が選択できます。
【FAQ】ChIP-qPCRのポジティブコントロールプライマー
H3K4me3は転写活性化に働くヒストンマークであるため、高いレベルで発現する遺伝子と相関していることが考えられます。
したがって、扱うサンプルで高いレベルの発現量を示している遺伝子領域をポジコン領域として、例えばGAPDHのようなハウスキーピング遺伝子などを使用できます。
それに対して、扱うサンプルで非活性な領域を選択する際は熟慮が必要ですが、そのような領域はネガコンとして使用できます。
また、ネガコンを設計する領域として、単純に活性なヒストンマークのない遺伝子間領域を選択することもできます。
【FAQ】ChIP-qPCRを複数のプレート上で行う場合
すべてのプレートで対照サンプルを配置し、対照の平均PCR効率に基づいてプレート間のCt値を正規化できます。
すべての計算の主な対照サンプルはインプットサンプルとし、一つのクロマチン調製物からのすべてのChIPおよびインプットサンプルを同じプレート上に配置します。
異なるターゲット(プライマーペア)を異なるプレートに配置して分割するようにしてください。
【FAQ】ChIP-qPCRのアンプリコンサイズについて
このアンプリコンのサイズはqPCRに最適なサイズ範囲からは外れますが、それでも機能する可能性はあります。
300bp未満のクロマチンフラグメントのうち、両方のプライマー配列を含み、増幅が可能な断片はごくわずかです。
小さいアンプリコンのプライマーペアを設計できない場合は、このような大きいアンプリコンが必要な場合があるかもしれません。
【FAQ】ChIP-qPCRに使用するDNA量
リアルタイムPCRには10~1000コピーの鋳型DNAが最適です。
これはゲノムDNAの場合は100 pg~1 μg、cDNAの場合は1 pg~100 ngのtotal RNAからの逆転写産物に相当します。
鋳型DNAが過剰の場合、不純物のレベルが高くなりPCR効率の低下につながります。